2024年诺贝尔物理学奖和化学奖双双授予ai领域,凸显了ai for science (ai4s) 的蓬勃发展,并预示着科学研究范式的变革。 信息工程学院田永鸿、陈杰团队长期致力于ai4s研究,其成果曾入围2022年度戈登贝尔特别奖,并荣获多项国家级和省级奖项。近日,该团队在《自然·机器智能》发表最新研究成果,再次证明ai在自然科学研究中的巨大潜力。
☞☞☞AI 智能聊天, 问答助手, AI 智能搜索, 免费无限量使用 DeepSeek R1 模型☜☜☜

论文发表界面截图
研究突破:
该研究针对病毒进化预测这一难题,提出了一种名为E2VD的进化驱动力预测框架。其亮点在于:
-
定制化蛋白质语言模型: 团队开发了针对进化预测任务定制的蛋白质语言模型,并设计了相应的预训练策略和数据集,优化了模型在预训练和下游任务之间的平衡。
-
跨病毒类型预测: E2VD模型能够预测多种病毒(包括新冠、流感、寨卡和艾滋病病毒)的进化轨迹,实现了跨病毒类型和跨毒株的通用预测。
-
突变相互作用网络重建: 模型包含一个突变相互作用网络重建模块,利用动态粒度注意力机制识别关键motif模式,并采用多任务焦点损失函数,提升了预测精度和模型的通用性。
-
多尺度进化预测: E2VD能够预测不同尺度的病毒进化趋势,未来可应用于疫苗和蛋白类药物设计,提高设计效率和可控性。
研究团队基于达尔文进化论和表观遗传学,从进化论视角出发,解决了病毒进化预测中“少数位点突变”和“稀少有益突变”两大难题。通过“微弱突变放大”和“稀少有益突变挖掘”策略,E2VD模型显著提升了预测精度,将稀少有益突变的预测精度从13%提升至80%。 模型在不同病毒类型和毒株上的泛化能力也得到了验证。
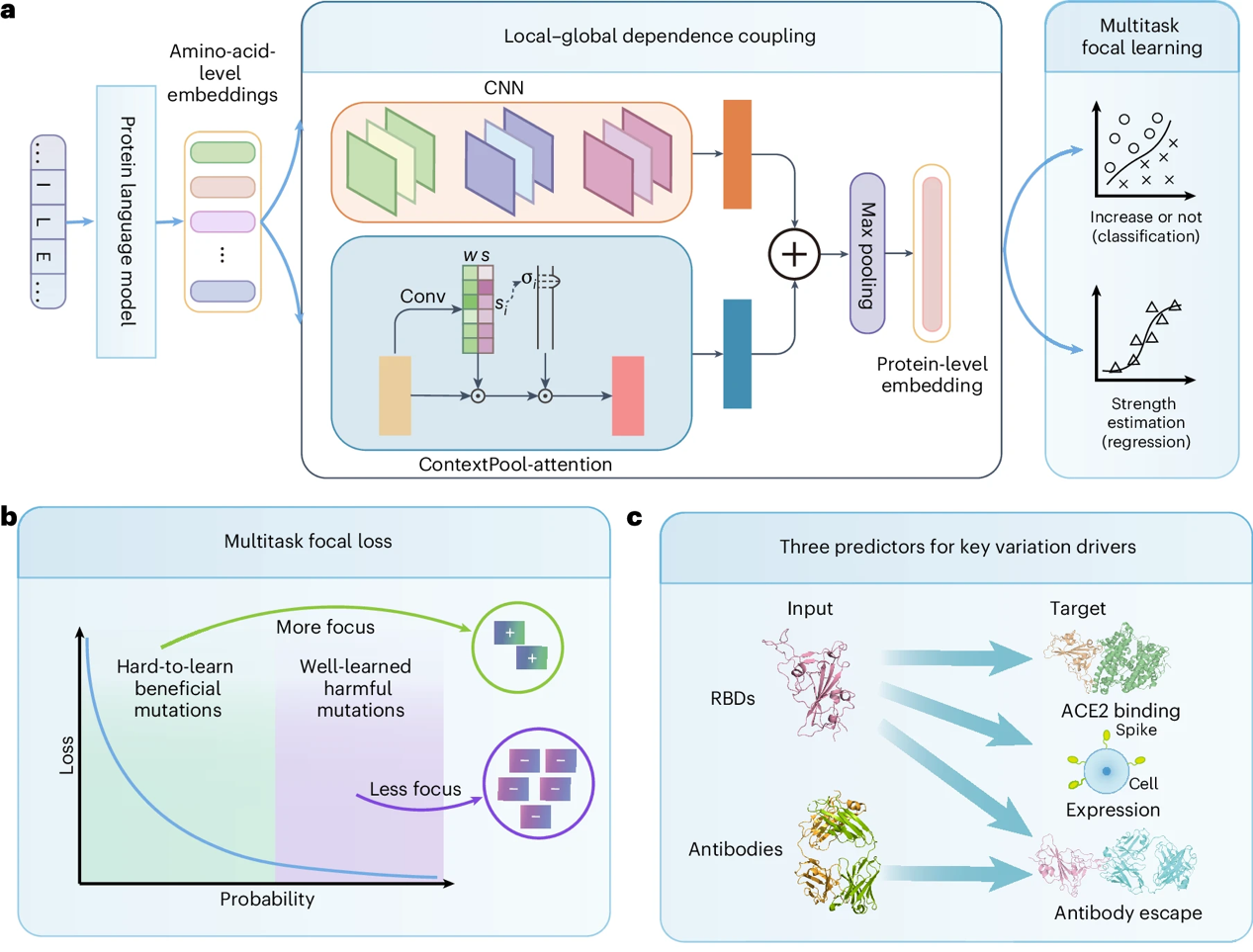
E2VD模型架构

跨病毒类型和跨毒株的泛化性能
该研究由北京大学信息工程学院田永鸿、陈杰团队完成,聂志伟和刘旭东为共同第一作者。 E2VD模型有望成为疫苗和药物研发的强大工具,加速对物种复杂进化机制的探索。






























